Molekularpathologie / Tumordiagnostik
Das Labor der Molekularpathologie verfügt mit der Next-Generation Sequencing-Technologie (NGS) über eine Plattform, die es erlaubt, Antworten auf sehr verschiedene Fragestellungen zu finden. Damit können Mutationen, Fusionen und Genamplifikationen sowie die Tumormutationslast detektiert werden. Wir sind ausserdem in der Lage, Therapie-induzierte Resistenzmutationen von Tumoren im peripheren Blut des Patienten zu finden und im Verlauf zu überwachen.
Wir bieten folgende NGS-Assays für Tumore an:
- KSBL Hotspot Profiler (HoP)
- Oncomine Comprehensive Assay (OCA)
- Oncomine Tumor Mutation Load (TML)
- Liquid Biopsy
Falls Sie Hilfe bei der Auswahl des richtigen Assays zu ihrer Fragestellung haben, können Sie sich jederzeit an uns wenden.
Molekularpathologie Einsendeformular (PDF)
Liquid Biopsy Einsendeformular (PDF)
Proben
Zur Frage nach relevanten Tumor- bzw. Driver-Mutationen, Genamplifikationen, Tumormutationslast oder Genfusionen können Sie gängiges pathologisches Untersuchungsmaterial einsenden, insbesondere Formalin-fixierte und Paraffin-eingebettete (FFPE) Proben, z.B. OP-Resektate, Biopsien und Feinnadelpunktionen.
Für die Detektion von Tumormutationen und Mutationen, die zu Therapieresistenz führen, senden Sie uns bitte 2 x 10mL peripheres Blut in zwei Cell-Free DNA BCT Röhrchen der Firma Streck.
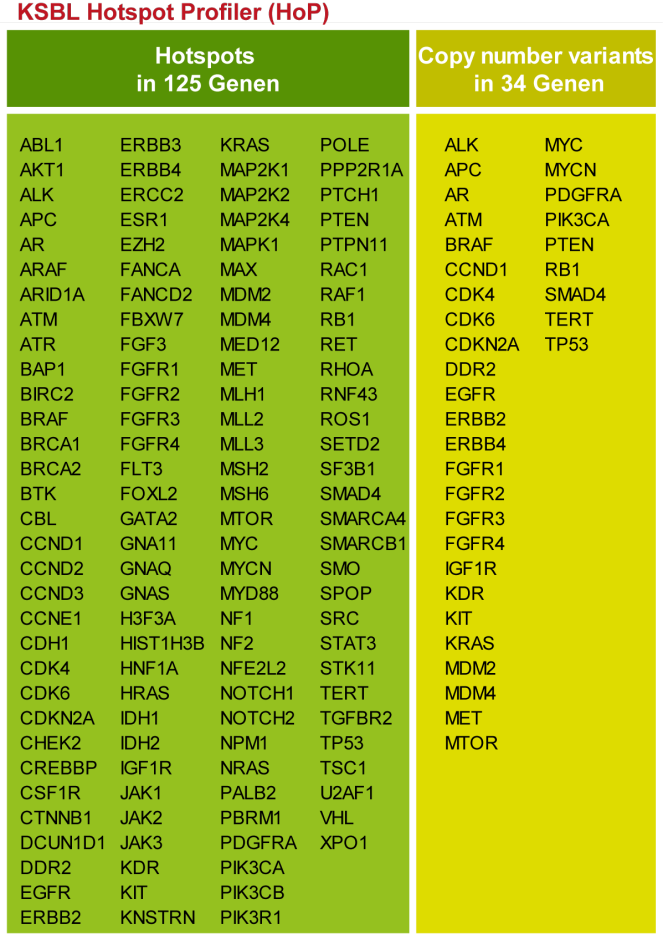
KSBL Hotspot Profiler (HoP)
Bei dem HoP handelt es sich um ein DNA-basiertes NGS-Panel, welches eigens vom Team der Molekularpathologie des KSBL entwickelt wurde. In einer einzigen Analyse des HoP werden dabei die DNA Genregionen von drei verschiedenen Assays abgedeckt: Oncomine Focus Assay, Ion AmpliSeq Cancer Hotspot Panel v2 und AmpliSeq™ Colon and Lung Cancer Research Panel v2. Ausserdem werden 80 Hotspot-Mutationen abgedeckt, die sonst nur im Oncomine Comprehensive Assays v3 enthalten wären
Mit dem HoP können bekannte Treibermutationen und Copy Number Variants (= «CNV», hauptsächlich Genamplifikationen) nachgewiesen werden.
Verwendung:
- Pan-Cancer Assay für besonders kleine Proben
- Frage nach Einzelmutationen (zB BRAF p.V600E zum Ausschluss von Lynch Syndrome)
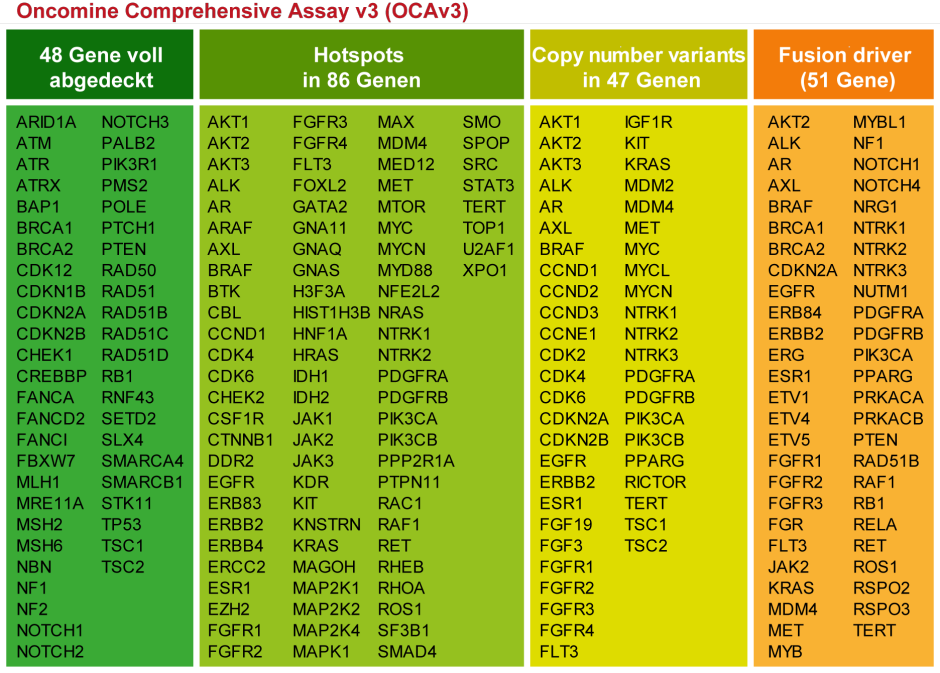
Oncomine Comprehensive Assay (OCA)
Der OCA ist ein mittelgrosses Pan-Cancer Panel. Es werden hier (im Gegensatz zum HoP) nicht nur Hotspots, sondern auch ausgewählte Gene komplett sequenziert (z.B. POLE, BRCA1 & BRCA2, …). Diese Gene sind zwar häufig mutiert, aber die Mutationen sammeln sich nicht in Hotspots. Damit eignet sich das Panel für komplexe Fälle oder solche, die im HoP bereits getestet wurden, ohne dass Mutationen gefunden wurden. Das Panel ist für Mamma- & Ovarialkarzinome gut geeignet, da sowohl Hotspots als auch einige DNA-Reparaturgene (BRCA1 & 2, RAD50/RAD 51 Familie, …) komplett abgedeckt werden.
Wie im HoP auch, sind Gene enthalten, die auf Copy Number Variants (= «CNV», hauptsächlich Genamplifikationen) hin untersucht werden.
Für die Detektion von Fusionen kann eine RNA Analyse angefordert werden. Aufgrund der Fragilität der RNA kann es (besonders bei kleinen Biopsien) passieren, dass nicht genügend RNA extrahiert werden kann, um die Fusionsanalyse erfolgreich durchzuführen.
Verwendung:
- Geeignet für Mamma- und Ovarialkarzinome (BRCA1/2 Analyse)
- Frage nach POLE-Mutationen wegen Immun-Checkpoint-Inhibitoren
- Frage nach Genfusionen
- komplexe Fälle, oder Fälle, welche im HoP keine therapierelevanten Treibermutationen gezeigt haben
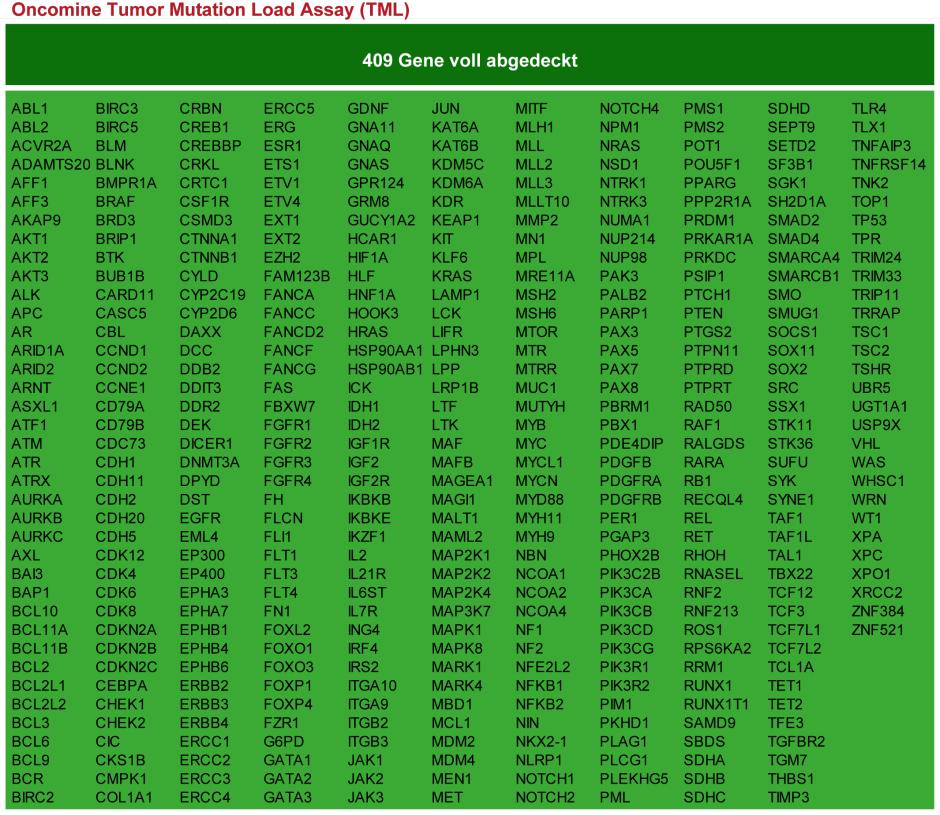
Oncomine Tumor Mutation Load (TML)
Der TML ist ein sehr grosses NGS Panel um die Anzahl somatischer Mutationen pro Megabase (1 Million Basenpaare) zu ermitteln. Das Ergebnis dieses grossen NGS-Assays ist die Tumormutationslast (TML), welche zur Abschätzung des Ansprechens auf eine Therapie mit Immun-Checkpoint-Inhibitoren benötigt wird.
Da der Test 409 sehr verschiedene Gene enthält, die ausnahmslos abgedeckt werden, kann der Assay aber auch für andere Fragestellungen nützlich sein. Zum Beispiel kann die Komplettabdeckung des VHL Gens hilfreich sein, um bei hellzelligen Nierenzellkarzinomen eine von Hippel-Lindau-Erkrankung zu diagnostizieren oder auszuschliessen. Ausserdem sind auch Gene enthalten, die z.B. bei hämatologischen Erkrankungen eine Rolle spielen (TET2, DNMT3A, SF3B1, etc). Dadurch bietet sich das Panel unter Umständen für Fälle an, die sich anderweitig nicht klar einordnen lassen.
In diesem Panel ist das POLE-Gen nicht enthalten (im OCAv3 voll abgedeckt). Auch bietet das Panel keine Möglichkeit, Copy Number Variants (= «CNV», hauptsächlich Genamplifikationen) oder Genfusionen zu detektieren.
Verwendung:
-
Zur Ermittlung der Mutationslast als prädiktiver Marker für eine Therapie mit Immun-Checkpoint-Inhibitoren
-
Nierenzellkarzinome (VHL)
- schwer klassifizierbare Tumoren.
Liquid Biopsy
Als nicht invasive Mutationsanalyse bieten wir Ihnen die Blut-basierte Analyse (sog. Liquid Biopsy) an, welche sich insbesondere im Rahmen der Verlaufskontrolle zur Frage nach Resistenzmutationen anbietet. Ein Indikator für das Auftreten einer Resistenzmutation kann die plötzliche Tumorprogression bei einem Patienten sein, der zuvor gut auf eine (zielgerichtete) Behandlung angesprochen hat. Hier kann die Liquid Biopsy den Verdacht auf eine Therapieresistenz des Tumors erhärten. Im besten Fall kann mit dem Wissen um die gefundene Resistenzmutation auf ein anderes, noch wirksameres Medikament umgestellt werden.
Bitte notieren Sie ihre Fragestellung und die allenfalls bereits bekannte(n) Tumormutation(en) auf dem Einsendeformular.
Liquid Biopsy Einsendeformular (PDF)
Die Assays sind besonders sensitiv und erlauben eine Detektion bis auf 0.1% Allelfrequenz (OFA bzw OCAv3: ca 10%). Daher eignen sie sich für die Detektion von Mutationen in zellfreier DNA oder von besonders kleinen Subklonen in soliden Tumoren. Die Sequenzierung von zellfreier DNA (cfDNA) von peripherem Blut bietet jedoch die Möglichkeit, bei schwer erreichbaren Tumoren (Lunge) ohne Biopsie, nach Driver- oder Resistenzmutationen zu suchen. Dafür sind bestimmte Blutentnahme-Röhrchen nötig. Hier am KSBL verwenden wir STRECK-Tubes (Cell-Free DNA BCT).
Aufgrund spezieller technischer Eigenschaften sind diese Assays sehr limitiert, was Anzahl und Grösse der untersuchten DNA-Regionen angeht. Daher sind die Assays auf den jeweiligen Tumor angepasst: Aktuell bieten wir spezifische Assays für Lungen- und Darmkrebs an. Ausserdem gibt es einen grösseren pan-Cancer Assay für komplexe Fälle und Forschungsfragen.
Verwendung
- Suche nach Resistenzmutationen im Blut
(z.B. bei Lungenkrebs Frage nach EGFR p.T790M oder p.C797x) - Monitoring der Driver-Mutation nach Behandlung (zB. im Rahmen von Studien des DKZ)
